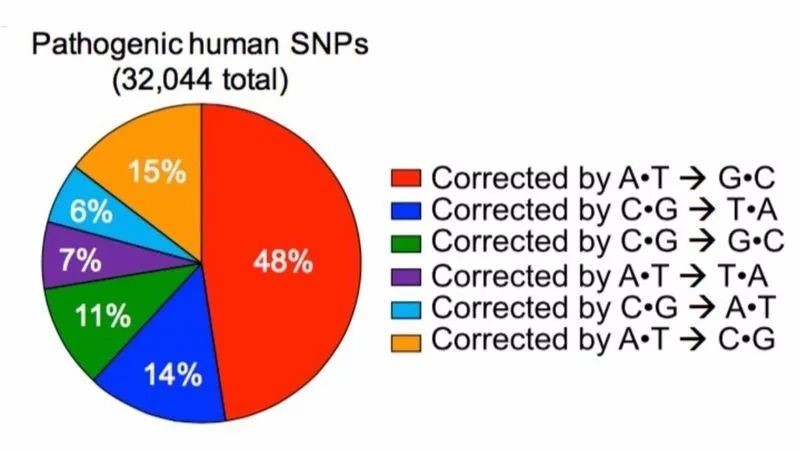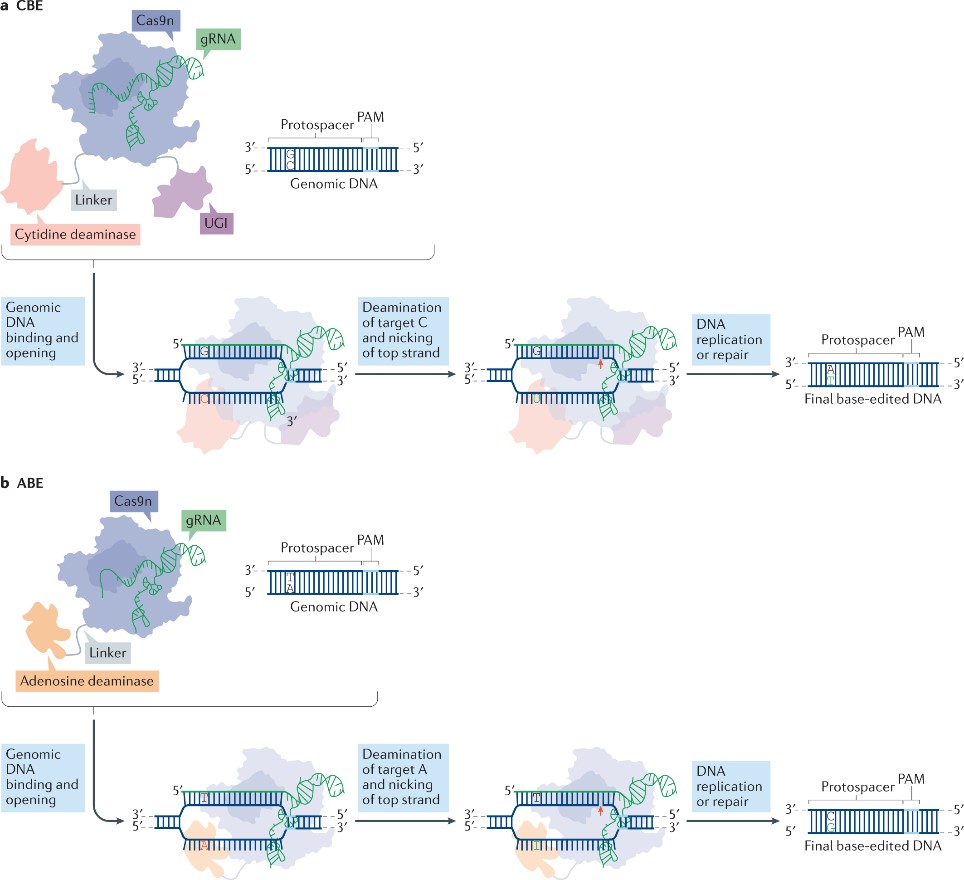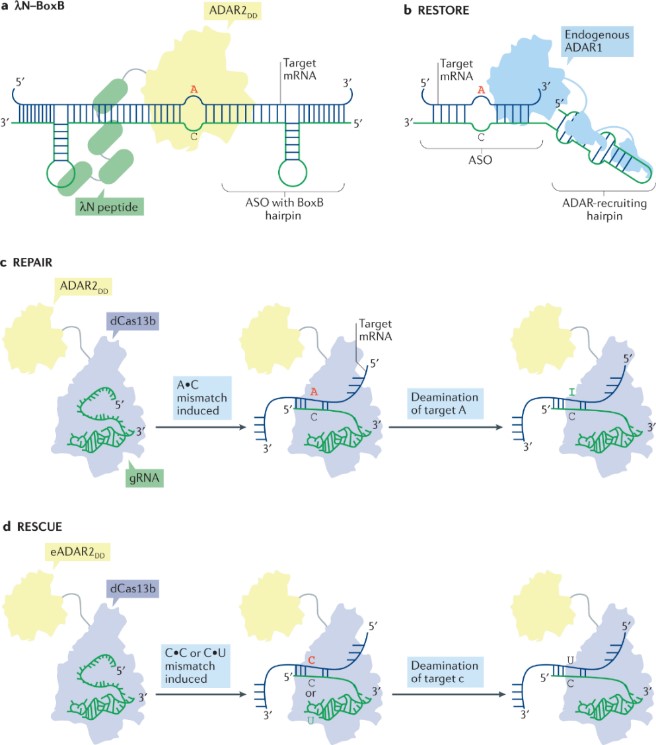
[5-8] a-c. A到I RNA碱基编辑;d. C到U RNA碱基编辑
- 人类疾病治疗的研究
2020年10月19日,加州大学欧文分校的研究人员在 Nature子刊 Nature Biomedical Engineering 杂志发表了题为:Restoration of visual function in adult mice with an inherited retinal disease via adenine base editing 的研究论文[9]。该研究使用慢病毒载体递送腺嘌呤单碱基编辑器(ABE),实现了对先天性黑蒙症小鼠模型突变基因的高效修复,有效恢复了小鼠模型视觉能力,且未发现可检测的脱靶效应。这项工作表明,单碱基编辑技术在某些情况下可能会替代基因增强疗法,以永久挽救因突变而失能的关键蛋白的功能,或修正无法使用基因增强疗法的显性遗传病。这项工作也代表了治疗遗传性视网膜疾病的新方向。
2020年10月19日,加州大学欧文分校的研究人员在 Nature子刊 Nature Biomedical Engineering 杂志发表了题为:Restoration of visual function in adult mice with an inherited retinal disease via adenine base editing 的研究论文[9]。该研究使用慢病毒载体递送腺嘌呤单碱基编辑器(ABE),实现了对先天性黑蒙症小鼠模型突变基因的高效修复,有效恢复了小鼠模型视觉能力,且未发现可检测的脱靶效应。这项工作表明,单碱基编辑技术在某些情况下可能会替代基因增强疗法,以永久挽救因突变而失能的关键蛋白的功能,或修正无法使用基因增强疗法的显性遗传病。这项工作也代表了治疗遗传性视网膜疾病的新方向。
- 动物模型开发
韩国首尔大学Jin-Soo Kim组率先发表了将 rAPOBEC-XTEN-Cas9n-UGI( BE3) 系统运用于小鼠疾病模型制备的研究成果[11]。针对小鼠抗肌萎缩蛋白基因Dmd设计sgRNA,通过显微注射小鼠受精卵,得到9只F0代小鼠中,有5只小鼠产生了靶位点的碱基突变,包括一只纯合子突变小鼠( CAG>TAG) ;针对小鼠酪氨酸酶基因Tyr设计sgRNA,通过电转染方法,得到7只F0后代小鼠中均检测到靶基因突变,包括3只纯合子Tyr突变小鼠。中山大学黄军就组也利用单碱基编 辑系统研究了该技术在制备小鼠疾病模型中的效率[12]。并首次报道了在小鼠胚胎中由碱基编辑系统诱导产生的靶位点处的碱基插入以及临近位点的脱氨基。
- 作物改良育种
中科院的高彩霞团队率先将单碱基编辑技术应用于三大重要农作物小麦、水稻和玉米的性状改良上,并且取得了重大进展[13]。高教授团队构建了密码子优化的 rAPOBEC1-XTEN-dCas9n-UGI 和 rAPOBEC1-XTEN-dCas9-UGI 两种融合蛋白,通过针 对三大农作物共 7 个基因位点进行单碱基编辑实验,发现选用 Cas9n 可以获得较高的单碱基编辑效率。同期中科院上海生命科学研究院的朱健康团队和中国农业科学院作物研究所的夏兰琴团队也分别报道了利用单碱基编辑系统对水稻基因进行单碱基编辑[14, 15]。单碱基编辑系统可以在农作物中取得高效的编辑效率,为改良作物品种带来新希望。[16]
-
Landrum, M. J. et al. ClinVar: public archive of interpretations of clinically relevant variants. Nucleic Acids Res. 44, D862–D868 (2016)
-
罗舒蕾. 单碱基编辑技术的研究进展[J]. 当代化工研究, 2020(5).
-
Komor, A. C., Kim, Y. B., Packer, M. S., Zuris, J. A. & Liu, D. R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature 533, 420–424 (2016).
-
Gaudelli, N. M. et al. Programmable base editing of A•T to G•C in genomic DNA without DNA cleavage. Nature 551, 464–471 (2017).
-
Montiel-Gonźalez, M. F., Vallecillo-Viejo, I. C. & Rosenthal, J. J. C. An efficient system for selectively altering genetic information within mRNAs. Nucleic Acids Res. 44, e157 (2016).
-
Merkle, T. et al. Precise RNA editing by recruiting endogenous ADARs with antisense oligonucleotides. Nat. Biotechnol. 37, 133–138 (2019).
-
Cox, D. B. T. et al. RNA editing with CRISPR–Cas13. Science 358, 1019–1027 (2017).
-
Abudayyeh, O. O. et al. A cytosine deaminase for programmable single-base RNA editing. Science 365, 382–386 (2019).
-
Suh, S., Choi, E.H., Leinonen, H. et al. Restoration of visual function in adult mice with an inherited retinal disease via adenine base editing. Nat Biomed Eng (2020).
-
Porto, E.M., Komor, A.C., Slaymaker, I.M. et al. Base editing: advances and therapeutic opportunities. Nat Rev Drug Discov (2020).
-
Porto, E.M., Komor, A.C., Slaymaker, I.M. et al. Base editing: advances and therapeutic opportunities. Nat Rev Drug Discov (2020).
-
Porto, E.M., Komor, A.C., Slaymaker, I.M. et al. Base editing: advances and therapeutic opportunities. Nat Rev Drug Discov (2020).
-
KIM K, RYU S M, KIM S T, et al. Highly efficient RNA-guided base editing in mouse embryos[J]. Nature Biotechnology,2017, 35 (5) : :435-437.
-
LIANG P, SUN H, SUN Y, et al. Effective gene editing by high-fidelity base editor 2 in mouse zygotes [J]. Protein & cell, 2017, DOI: 10. 1007 /s13238-017-0418-2
-
ZONG Y, WANG Y, LI C, et al. Precise base editing in rice,wheat and maize with a Cas9-cytidine deaminase fusion [J]. Nature Biotechnology, 2017, 35 (5): 438-440.
-
LU Y M, ZHU J K. Precise Editing of a Target Base in the Rice Genome Using a Modified CRISPR/Cas9 System [J]. Molecular plant, 2017,10 (3): 523-525.
-
LI J, SUN Y, DU J, et al. Generation of Targeted Point Mutations in Rice by a Modified CRISPR/Cas9 System [J]. Molecular plant, 2017, 10 ( 3) : 526-529.
-
刘佳慧, 梁普平, 时光, et al. 单碱基基因编辑系统的研究进展 [J]. 世界科技研究与发展, 2017, 039(006):457-462.



























 David R. Liu
David R. Liu